AlphaFold
| Makale serisidir |
| Yapay zekâ |
|---|
AlphaFold, Alphabet'in bir yan kuruluşu olan DeepMind tarafından protein yapısını tahmin etme üzerine geliştirilen bir yapay zeka programı.[1] Program, bir derin öğrenme sistemi olarak tasarlanmıştır.[2]
AlphaFold yazılımının üç ana versiyonu bulunmaktadır. AlphaFold 1 (2018) kullanan bir araştırma ekibi, Aralık 2018'de 13. CASP genel sıralamasında birinci oldu. Program, yarışma organizatörleri tarafından en zor olarak değerlendirilen ve kısmen benzer diziye sahip proteinlerden mevcut şablon yapıları bulunmayan hedefler için en doğru yapıyı tahmin etmede özellikle başarılı oldu. AlphaFold 2 (2020) kullanan bir ekip, Kasım 2020'deki CASP14 yarışmasında bu konumunu tekrarladı.[3] Ekip, diğer gruplardan çok daha yüksek bir doğruluk seviyesine ulaştı.[2][4] CASP'ın global mesafe testi (GDT) adı verilen ve bir hesaplama programının tahmin ettiği yapının laboratuvarda belirlenen yapıya ne kadar benzediğini ölçen testte, 100 tam bir eşleşme olacak şekilde, iki protein arasındaki mesafe eşiği kullanılarak hesaplanan GDT'de, proteinlerin yaklaşık üçte ikisi için 90'ın üzerinde bir skor elde etti.[2][5]
AlphaFold 2'nin CASP14'teki sonuçları "şaşırtıcı"[6] ve "dönüştürücü" olarak tanımlandı.[7] Bazı araştırmacılar, tahminlerinin üçte biri için doğruluğun yeterince yüksek olmadığını ve protein katlanması sorununu çözmek için mekanizmayı veya kuralları ortaya koymadığını belirtti.[8][9] Buna rağmen, teknik başarı geniş çapta takdir gördü. 15 Temmuz 2021'de AlphaFold 2 makalesi, Nature'da ön erişim yayını olarak, açık kaynak yazılım ve türlerin proteomlarının aranabilir bir veritabanı ile birlikte olarak yayımlandı.[10][11][12]
AlphaFold 3, 8 Mayıs 2024'te duyuruldu. Bu versiyon, DNA, RNA, çeşitli ligandlar ve iyonlar ile proteinler tarafından oluşturulan protein komplekslerinin yapısını tahmin edebilmektedir.
Arka Plan

Proteinler, kendiliğinden katlanarak proteinlerin üç boyutlu (3D) yapısını oluşturan amino asit zincirlerinden oluşur. 3D yapı, proteinin biyolojik fonksiyonunu anlamak için hayati öneme sahiptir.
Protein yapıları, X ışını kristalografisi, krio-elektron mikroskopisi ve nükleer manyetik rezonans gibi deneysel teknikler kullanılarak belirlenebilir, ancak bu teknikler pahalı ve zaman alıcıdır. Bu tür çabalar, deneysel yöntemler kullanarak son 60 yılda yaklaşık 170.000 proteinin yapısını belirlemişken, tüm yaşam formlarında bilinen protein sayısı 200 milyondan fazladır.[5]
Yıllar boyunca, araştırmacılar amino asit dizilerinden proteinlerin 3D yapılarını tahmin etmek için birçok hesaplama yöntemini uygulamışlardır, ancak bu yöntemlerin doğruluğu deneysel tekniklere yakın olmamıştır. 1994 yılında bilim topluluğunu en iyi protein yapı tahminlerini üretmeye zorlamak amacıyla başlatılan CASP, 2016 yılına kadar en zor proteinler için sadece yaklaşık 40/100 GDT skorlarının elde edilebileceğini bulabilmişti.[5] AlphaFold, 2018 CASP'ta bir yapay zeka (AI) derin öğrenme tekniği kullanarak bu yarışmaya başladı.
Algoritma
DeepMind'in, protein dizileri ve yapılarından oluşan halka açık bir depodan 170.000'den fazla protein üzerinde programı eğittiği bilinmektedir. Program, dikkat ağı olarak bilinen bir derin öğrenme tekniği kullanmaktadır. Bu teknik, yapay zekâ'nın daha büyük bir sorunun parçalarını tanımlayıp ardından genel çözümü elde etmek için bu parçaları bir araya getirmesine odaklanmaktadır.[2] Genel eğitim, 100 ile 200 arasında GPU işlem gücü üzerinde gerçekleştirilmiştir.[2]
AlphaFold 1 (2018)
AlphaFold 1 (2018), 2010'larda çeşitli ekipler tarafından geliştirilen çalışmalara dayanıyordu. Bu çalışmalar, şimdi birçok farklı organizmadan elde edilen ilgili DNA dizilerinin büyük veri bankalarına bakarak, ana zincirde ardışık olmasalar bile, farklı kalıntılarda ortaya çıkan değişikliklerin ilişkili olduğunu bulmaya çalışıyordu. Bu tür korelasyonlar, kalıntıların dizide birbirine yakın olmasalar bile fiziksel olarak birbirine yakın olabileceğini düşündürmektedir, bu da bir temas haritasının tahmin edilmesini sağlar. 2018 öncesindeki son çalışmalara dayanarak, AlphaFold 1 bunu genişleterek kalıntıların ne kadar yakın olma olasılığının tahmin edilmesi için bir olasılık dağılımı tahmin etti—temas haritasını olası bir mesafe haritasına dönüştürdü. Ayrıca, çıkarım geliştirmek için önceki yöntemlere göre daha gelişmiş öğrenme yöntemleri kullandı.[13][14]
AlphaFold 2 (2020)
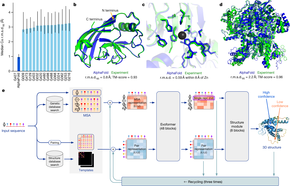

DeepMind ekibine göre, 2020 versiyonu, 2018'de CASP 13'ü kazanan orijinal versiyondan önemli ölçüde farklıdır.[16][17]
AlphaFold 1'de kullanılan yazılım tasarımı, her biri ayrı ayrı eğitilen ve daha sonra fizik tabanlı enerji potansiyeli ile birleştirilen kılavuz potansiyeli üretmek için kullanılan bir dizi modül içeriyordu. AlphaFold 2, bunun yerine tamamen desen tanıma üzerine kurulu, tek bir farklılaştırılabilir uçtan uca modele birleştirilmiş alt ağlar sistemini getirdi ve bu, tek bir entegre yapı olarak bütünleşik bir şekilde eğitildi.[17][18] Yerel fizik, AMBER modeline dayalı enerji iyileştirme şeklinde, yalnızca sinir ağı tahmini birleştikten sonra son bir iyileştirme adımı olarak uygulanır ve tahmin edilen yapıyı sadece biraz ayarlar.[19]
2020 sisteminin önemli bir parçası, her biri bir proteinin amino asit kalıntısı ile başka bir amino asit kalıntısı arasındaki ilişki (graf teori terminolojisinde "kenar") için bir bilgi vektörünü aşamalı olarak iyileştirmek için kullanılan, bir Transformer tasarımına dayandığı düşünülen iki modüldür (bu ilişkiler yeşil renkte gösterilen dizi ile temsil edilir); ve her amino asit pozisyonu ile giriş dizi hizalamasındaki her farklı diziler arasındaki ilişkiler (bu ilişkiler kırmızı renkte gösterilen dizi ile temsil edilir).[18] İçsel olarak, bu iyileştirme dönüşümleri, bu ilişkiler için bağlam bağımlı bir şekilde, eğitim verilerinden öğrenilen, ilgili verileri bir araya getirmenin ve ilgisiz verileri filtrelemenin ("dikkat mekanizması") etkisini içeren katmanlar içerir. Bu dönüşümler yinelenir, bir adımın güncellenmiş bilgi çıktısı bir sonrakinin girdisi olur, keskinleşen kalıntı/kalıntı bilgisi, kalıntı/dizi bilgisinin güncellenmesine katkıda bulunup daha sonrasında iyileşen kalıntı/dizi bilgisi, kalıntı/kalıntı bilgisinin güncellenmesine katkıda bulunur.[18] Bir rapora göre yineleme ilerledikçe, "dikkat algoritması ... bir kişinin bir yapbozu birleştirme biçimini taklit eder: önce küçük kümelerdeki parçaları—bu durumda amino asit kümeleri—birbirine bağlayarak ve ardından kümeleri daha büyük bir bütün halinde birleştirmenin yollarını arayarak."[5]
Bu yinelemelerin çıktısı, nihai yapı tahmin modülünü bilgilendirir,[18] ki bu modül de Transformerlar kullanır,[20] ve kendisi de yinelemelidir. DeepMind tarafından sunulan bir örnekte, yapı tahmin modülü ilk yinelemesinde hedef protein için doğru bir topoloji elde etti ve GDT_TS skoru 78 olarak değerlendirildi, ancak büyük bir sayıda (90%) stereokimyasal ihlal – yani fiziksel olmayan bağ açıları veya uzunlukları – içeriyordu. Sonraki yinelemelerle stereokimyasal ihlallerin sayısı azaldı. Üçüncü yinelemede tahminin GDT_TS skoru 90'a yaklaşıyor ve sekizinci yinelemede stereokimyasal ihlaller neredeyse sıfıra iniyordu.[21]
Eğitim verileri başlangıçta tek peptid zincirleri ile sınırlıydı. Ancak, Ekim 2021'de yayınlanan ve AlphaFold-Multimer olarak adlandırılan güncelleme, eğitim verilerine protein komplekslerini dahil etti. DeepMind, bu güncellemenin protein-protein etkileşimlerini doğru bir şekilde tahmin etmede yaklaşık %70 başarı sağladığını belirtti.[22]
AlphaFold 3 (2024)
8 Mayıs 2024'te duyurulan AlphaFold 3, Alphabet'in yan kuruluşları olan Google DeepMind ve Isomorphic Labs tarafından ortaklaşa geliştirildi. AlphaFold 3, tek zincirli proteinlerle sınırlı olmayıp, DNA, RNA, sonrası translasyonel modifikasyonlar ve belirli ligandlar ve iyonlar ile protein komplekslerinin yapılarını da tahmin edebilmektedir.[23]
AlphaFold 3, AlphaFold 2 ile tanıtılan Evoformer'a benzer ancak daha basit bir yapı olan "Pairformer" adlı derin öğrenme mimarisini tanıtmıştır.[24][25] Pairformer modülünden gelen ham tahminler bir difüzyon modeline aktarılmakta, bu model atom bulutuyla başlayıp bu tahminleri kullanarak moleküler yapının 3 boyutlu bir tasvirine doğru iteratif olarak ilerlemektedir.
AlphaFold sunucusu, ticari olmayan araştırmalar için AlphaFold 3'e ücretsiz erişim sağlamak amacıyla erişime açıktır.[26]
Kaynak kodu
DeepMind, bilim camiasından gelen talepler üzerine AlphaFold'un çeşitli sürümlerinin (AlphaFold 3 hariç) kaynak koduna açık erişim sağlamıştır.[27][28][29] AlphaFold-3'ün tam kaynak kodunun 2024 yılı sonuna kadar açık erişime sunulması beklenmektedir.[30][31]
Uygulamalar
AlphaFold, COVID-19'un neden olduğu SARS-CoV-2 virüsünün proteinlerinin yapılarının tahmininde kullanılmıştır. Bu proteinlerin yapıları, 2020'nin başlarında deneysel olarak belirlenmekteydi.[7][32] Sonuçlar, geniş araştırma topluluğuna sunulmadan önce Birleşik Krallık'taki Francis Crick Institute'teki bilim insanları tarafından incelenmiştir. Ekip ayrıca, hesaplamalı olarak belirlenen yapıların Protein Veri Bankası'nda (PDB) paylaşılan deneysel olarak belirlenen SARS-CoV-2 spike proteini ile karşılaştırıldığını doğrulamıştır.[33] Ekip, bu protein yapıların devam eden terapötik araştırma çabalarının konusu olmasa da, SARS-CoV-2 virüsünün anlaşılmasına katkıda bulunacağını kabul etmiştir.[33] Özellikle, AlphaFold 2'nin ORF3a proteininin yapısının tahmini, kriyo-elektron mikroskopisi kullanarak University of California, Berkeley'deki araştırmacılar tarafından belirlenen yapıya çok benzemektedir. Bu spesifik proteinin, virüsün konak hücreden çıkmasına yardımcı olduğu ve enfeksiyona karşı inflamatuar yanıtı tetiklemekte rol oynadığı düşünülmektedir.[34]
Kaynaklar
- ^ "AlphaFold". Deepmind. 19 Ocak 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 30 Kasım 2020.
- ^ a b c d e "DeepMind's protein-folding AI has solved a 50-year-old grand challenge of biology". MIT Technology Review (İngilizce). 28 Ağustos 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 30 Kasım 2020.
- ^ Shead, Sam (30 Kasım 2020). "DeepMind solves 50-year-old 'grand challenge' with protein folding A.I." CNBC (İngilizce). 28 Ocak 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 30 Kasım 2020.
- ^ Stoddart, Charlotte (1 Mart 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. 7 Nisan 2022 tarihinde kaynağından arşivlendi. Erişim tarihi: 25 Mart 2022.
- ^ a b c d Robert F. Service, 'The game has changed.' AI triumphs at solving protein structures 24 Haziran 2023 tarihinde Wayback Machine sitesinde arşivlendi., Science, 30 November 2020
- ^ Mohammed AlQuraishi, CASP14 scores just came out and they're astounding 4 Ağustos 2022 tarihinde Wayback Machine sitesinde arşivlendi., Twitter, 30 November 2020.
- ^ a b Callaway, Ewen (30 Kasım 2020). "'It will change everything': DeepMind's AI makes gigantic leap in solving protein structures". Nature (İngilizce). 588 (7837). ss. 203-204. Bibcode:2020Natur.588..203C. doi:10.1038/d41586-020-03348-4. PMID 33257889.
- ^ Stephen Curry, No, DeepMind has not solved protein folding 29 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi., Reciprocal Space (blog), 2 December 2020
- ^ Balls, Phillip (9 Aralık 2020). "Behind the screens of AlphaFold". Chemistry World. 15 Ağustos 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 8 Ağustos 2024.
- ^ Jumper, John; Evans, Richard; Pritzel, Alexander; Green, Tim; Figurnov, Michael; Ronneberger, Olaf; Tunyasuvunakool, Kathryn; Bates, Russ; Žídek, Augustin; Potapenko, Anna; Bridgland, Alex; Meyer, Clemens; Kohl, Simon A A; Ballard, Andrew J; Cowie, Andrew; Romera-Paredes, Bernardino; Nikolov, Stanislav; Jain, Rishub; Adler, Jonas; Back, Trevor; Petersen, Stig; Reiman, David; Clancy, Ellen; Zielinski, Michal; Steinegger, Martin; Pacholska, Michalina; Berghammer, Tamas; Bodenstein, Sebastian; Silver, David; Vinyals, Oriol; Senior, Andrew W; Kavukcuoglu, Koray; Kohli, Pushmeet; Hassabis, Demis (15 Temmuz 2021). "Highly accurate protein structure prediction with AlphaFold". Nature (İngilizce). 596 (7873). ss. 583-589. Bibcode:2021Natur.596..583J. doi:10.1038/s41586-021-03819-2. PMC 8371605 $2. PMID 34265844.
- ^ "GitHub - deepmind/alphafold: Open source code for AlphaFold". GitHub (İngilizce). 23 Temmuz 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 24 Temmuz 2021.
- ^ "AlphaFold Protein Structure Database". alphafold.ebi.ac.uk. 24 Temmuz 2021 tarihinde kaynağından arşivlendi. Erişim tarihi: 24 Temmuz 2021.
- ^ Mohammed AlQuraishi (May 2019), AlphaFold at CASP13 22 Kasım 2021 tarihinde Wayback Machine sitesinde arşivlendi., Bioinformatics, 35(22), 4862–4865 DOI:10.1093/bioinformatics/btz422. See also Mohammed AlQuraishi (December 9, 2018), AlphaFold @ CASP13: "What just happened?" 29 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi. (blog post).
Mohammed AlQuraishi (15 January 2020), A watershed moment for protein structure prediction 23 Haziran 2022 tarihinde Wayback Machine sitesinde arşivlendi., Nature 577, 627–628 DOI:10.1038/d41586-019-03951-0 - ^ AlphaFold: Machine learning for protein structure prediction 12 Mayıs 2022 tarihinde Wayback Machine sitesinde arşivlendi., Foldit, 31 January 2020
- ^ a b Jumper, John; ve diğerleri. (August 2021). "Highly accurate protein structure prediction with AlphaFold". Nature (İngilizce). 596 (7873). ss. 583-589. Bibcode:2021Natur.596..583J. doi:10.1038/s41586-021-03819-2. ISSN 1476-4687. PMC 8371605 $2. PMID 34265844.
- ^ "DeepMind is answering one of biology's biggest challenges". The Economist. 30 Kasım 2020. ISSN 0013-0613. 3 Aralık 2020 tarihinde kaynağından arşivlendi. Erişim tarihi: 30 Kasım 2020.
- ^ a b Jeremy Kahn, DeepMind'ın protein katlanması üzerindeki yapay zeka atılımından çıkarılan dersler 8 Nisan 2022 tarihinde Wayback Machine sitesinde arşivlendi., Fortune, 1 Aralık 2020
- ^ a b c d Bknz. blok diyagramı. Ayrıca John Jumper et al. (1 Aralık 2020), AlphaFold 2 sunumu 3 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi., slayt 10
- ^ John Jumper ve diğerleri, konferans özeti (Aralık 2020)
- ^ Yapı modülünün "3-boyutlu eşdeğer dönüştürücü mimarisi" kullandığı belirtilmiştir (John Jumper et al. (1 Aralık 2020), AlphaFold 2 sunumu 3 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi., slayt 12).
SE(3)-eşdeğerlik ile bir dönüştürücü ağ tasarımı, Fabian Fuchs et al SE(3)-Dönüştürücüler: 3D Roto-Çeviri Eşdeğer Dikkat Ağları 7 Ekim 2021 tarihinde Wayback Machine sitesinde arşivlendi., NeurIPS 2020'de önerildi; ayrıca web sitesi 3 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi.. Bunun AlphaFold'da kullanılanla ne kadar benzer ya da farklı olduğu bilinmemektedir.
AlQuaraishi'nin bu konudaki blog yazısı 8 Aralık 2020 tarihinde Wayback Machine sitesinde arşivlendi. veya Fabian Fuchs'un daha detaylı yazısı 3 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi. incelenebilir - ^ John Jumper et al. (1 Aralık 2020), AlphaFold 2 sunumu 3 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi., slaytlar 12-20
- ^ Callaway, Ewen (13 Nisan 2022). "What's next for AlphaFold and the AI protein-folding revolution". Nature (İngilizce). 604 (7905). ss. 234-238. Bibcode:2022Natur.604..234C. doi:10.1038/d41586-022-00997-5. PMID 35418629.
- ^ Metz, Cade (8 Mayıs 2024). "Google Unveils A.I. for Predicting Behavior of Human Molecules". The New York Times (İngilizce). ISSN 0362-4331. Erişim tarihi: 9 Mayıs 2024.
- ^ Abramson, Josh; Adler, Jonas; Dunger, Jack; Evans, Richard; Green, Tim; Pritzel, Alexander; Ronneberger, Olaf; Willmore, Lindsay; Ballard, Andrew J.; Bambrick, Joshua; Bodenstein, Sebastian W.; Evans, David A.; Hung, Chia-Chun; O’Neill, Michael; Reiman, David (8 Mayıs 2024). "Accurate structure prediction of biomolecular interactions with AlphaFold 3". Nature (İngilizce). ss. 1-3. doi:10.1038/s41586-024-07487-w. ISSN 1476-4687. PMC 11168924 $2. 17 Mayıs 2024 tarihinde kaynağından arşivlendi. Erişim tarihi: 8 Ağustos 2024.
- ^ Accurate structure prediction of biomolecular interactions with AlphaFold 3 12 Mayıs 2024 tarihinde Wayback Machine sitesinde arşivlendi., pdf of preprint of the article in Nature.
- ^ A non-commercial server of AlphaFold-3
- ^ Domínguez, Nuño (2 Aralık 2020). "La inteligencia artificial arrasa en uno de los problemas más importantes de la biología". El País (İspanyolca). 26 Temmuz 2022 tarihinde kaynağından arşivlendi. Erişim tarihi: 12 Mayıs 2024.
- ^ Briggs, David (4 Aralık 2020). "If Google's Alphafold2 really has solved the protein folding problem, they need to show their working". The Skeptic (İngilizce). 12 Mayıs 2024 tarihinde kaynağından arşivlendi. Erişim tarihi: 12 Mayıs 2024.
- ^ Demis Hassabis, "Brief update on some exciting progress on #AlphaFold!" 22 Temmuz 2022 tarihinde Wayback Machine sitesinde arşivlendi. (tweet), via twitter, 18 June 2021
- ^ "AlphaFold3 — why did Nature publish it without its code?". 28 Haziran 2024 tarihinde kaynağından arşivlendi. Erişim tarihi: 8 Ağustos 2024.
- ^ "AlphaFold 3: Stepping into the future of structure prediction". 27 Mayıs 2024 tarihinde kaynağından arşivlendi. Erişim tarihi: 8 Ağustos 2024.
- ^ "AI Can Help Scientists Find a Covid-19 Vaccine". Wired (İngilizce). ISSN 1059-1028. 23 Nisan 2022 tarihinde kaynağından arşivlendi. Erişim tarihi: 1 Aralık 2020.
- ^ a b "Computational predictions of protein structures associated with COVID-19". Deepmind. 4 Ağustos 2020. 25 Mart 2022 tarihinde kaynağından arşivlendi. Erişim tarihi: 1 Aralık 2020.
- ^ "How DeepMind's new protein-folding A.I. is already helping to combat the coronavirus pandemic." Fortune (İngilizce). 30 Kasım 2020 tarihinde kaynağından arşivlendi. Erişim tarihi: 1 Aralık 2020.